Dans le cadre de travaux visant à sécuriser la filière française de réactifs d'immuno-diagnostic pour la détection et l'identification du virus SARS-CoV-2, le Laboratoire Innovations technologiques pour la Détection et le Diagnostic (SPI/Marcoule) a été chargé de produire du virus inactivé dans ses installations de sécurité biologique de niveau 3 (L3) à Bagnols-sur-Cèze afin de créer de nouveaux anticorps monoclonaux. Les équipes du Li2D se sont ainsi mobilisées pour, dans un premier temps, optimiser les conditions d'amplification virale dans des cellules en culture et obtenir du matériel viral de qualité qui a été analysé par spectrométrie de masse. La spectrométrie de masse est une méthode de choix, car elle permet une analyse rapide et fiable des protéines du virus en milieu complexe (lien vers l'actualité correspondante), mais elle a également renseigné sur les évènements moléculaires se produisant suite à l'infection d'une cellule par le virus, objet de la présente étude.
Dans cette étude, accomplie en un temps record, les chercheurs ont analysé la dynamique du protéome de cellules Vero E6 infectées par la souche italienne du virus SRAS-CoV-2 à deux multiplicités d'infection (ratio virus/cellules) pendant 7 jours (Figure). Les protéines du virus SARS-CoV-2 sont synthétisées de façon continue par les cellules avec une production maximale enregistrée à jour J3 post-infection dans les conditions expérimentales testées. La technologie de dernière génération utilisée* a permis d'identifier et de quantifier un total de 3220 protéines de l'hôte. L'abondance de ces protéines suivies sur plusieurs jours a montré que certains processus biologiques de l'hôte étaient affectés par le virus. Par exemple, des composants du spliceosome, de la maturation des protéines dans le réticulum endoplasmique ou de la formation de vacuoles sont autant de pistes d'intérêt révélées par l'analyse de réseaux d'interactions fonctionnelles. Ces résultats, publiés dans Emerging Microbes & Infections, ont été dévoilés en avant-première sur BioRxiv, archive de dépôt de préprints, afin de partager au plus vite les informations avec la communauté scientifique dans l'espoir d'accélérer le développement de contremesures thérapeutiques et de rapidement progresser sur la compréhension du fonctionnement de ce nouveau virus.
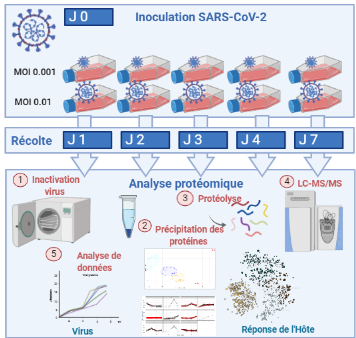
Schéma de la méthodologie employée
Actualité adaptée du texte publié par le département SDV de l'Université Paris-Saclay http://sco.lt/6gyi4e
* La protéomique « shotgun » fait référence à l'utilisation de techniques de protéomique pour identifier les protéines dans des mélanges complexes en utilisant une combinaison de chromatographie liquide à haute performance combinée à la spectrométrie de masse.