Parce que le centre Neurospin est dirigé par l'inventeur de l'imagerie de diffusion, l'équipe METRIC est experte dans ce domaine et travaille activement à son développement. L'imagerie de diffusion est devenue populaire depuis deux décennies car elle reste la seule technique d'accès in vivo à la connectivité anatomique du cerveau humain. L'imagerie du processus de diffusion de l'eau dans le cerveau repose sur l'observation statistique du libre déplacement des molécules d'eau dans le tissu qui est entravé ou restreint par les membranes des cellules qui le composent. Ce mouvement est particulièrement restreint au sein de a substance blanche constituée pour l'essentiel des fibres axonales entourées d'une gaine protectrice de myéline qui assure l'efficacité du transfert de l'influx nerveux. Ces fibres sont disposées parallèlement et ajencées en faisceaux plus ou moins gros qui constituent les « grandes autoroutes » de l'information entre les aires fonctionnelles à la surface du cortex et les structures profondes telles que les noyaux gris centraux. Cette organisation très anisotrope de la substance blanche peut être inférée en observant par IRM le processus de diffusion en son sein qui est très restreint dans la direction principale des fibres. Les trajectoires des fibres peuvent alors être reconstruites grâce à des techniques dites de tractographies qui reposent sur la connaissance en tout point du cerveau de la distribution des orientations du processus de diffusion.
L'équipe a développé un savoir-faire tant sur le volet du développement de séquences innovantes d'acquisition que sur le volet de l'analyse, et est partenaire du consortium européen CONNECT rassemblant les 10 équipes à la pointe dans ce domaine travaillant à l'étude du connectome humain.
Dans ce cadre, elle a développé la plateforme logicielle Connectomist intégrant toutes les étapes nécessaires à l'étude du connectome structurel, et a publié le premier atlas de la connectivité anatomique cérébrale, comme l'illustre la figure suivante.
 |  |
Au delà du profil angulaire des déplacements des molécules d'eau, l'IRMd permet véritablement de sonder le tissu l'échelle cellulaire parce qu'au cours de leur mouvement d'amplitude microscopique, les molécules d'eau rencontrent les parois des cellules (gliales, neuronales, axonales, astrocytaires, ….) et encodent donc la géométrie membranaire de ces cellules. Tout le travail du biophysicien consiste alors à “décoder” cette information en modélisant les différents tissus cérébraux à l'aide de modèles mathématiques simples de cette géométrie membranaire. C'est à la fin des années 2000 qu'est apparue cette nouvelle modalité en voie de transformer l'IRM en un fantastique outil de biopsie virtuelle permettant l'exploration non invasive de l'organisation des tissus à l'échelle cellulaire : la microscopie par IRM de diffusion ou μIRMd . Le centre NeuroSpin a réalisé les développements méthodologiques en IRM permettant aujourd'hui d'offrir aux utilisateurs de la plateforme de NeuroSpin les séquences de microscopie par IRM de diffusion (Lebois et al, 2012) ainsi que les outils d'analyse associés dont nous avons pu démontrer l'efficacité en développant le premier atlas quantitatif de la microstructure des grands faisceaux de la substance, comme illustré en figure 4 (Assaf et al, 2013 ; Duclap et al, 2013 , Schmitt et al, 2012).
L’imageur clinique 7T du centre NeuroSpin est doté d’un système de gradients très puissant capable de délivrer 80mT/m en plus de disposer d'un champ magnétique unique en France aujourd'hui, permettant de développer des protocoles de microscopie de diffusion bien plus performant qu'à 3T, grâce notamment à la possibilité offerte par cette séquence de régler indépendamment la forme des impulsions et le temps de diffusion. L'on peut ainsi programmer des gradients trapézoidaux à l’instar du schéma classique développé par Stejskal et Tanner (Pulsed Gradient Spin Echo ou PGSE) et utilisé dans les protocoles AxCaliber et ActiveAx, mais également des gradients sinusoidaux (Oscillating Gradient Spin Echo ou OGSE) comme proposé par Does (Does et al, 2003), ou encore des formes de gradients arbitraires (Arbitrary Spin Echo ou ASE) comme propose par Callaghan et repris récemment par Drobjnak (Drobjnak et al, 2011).
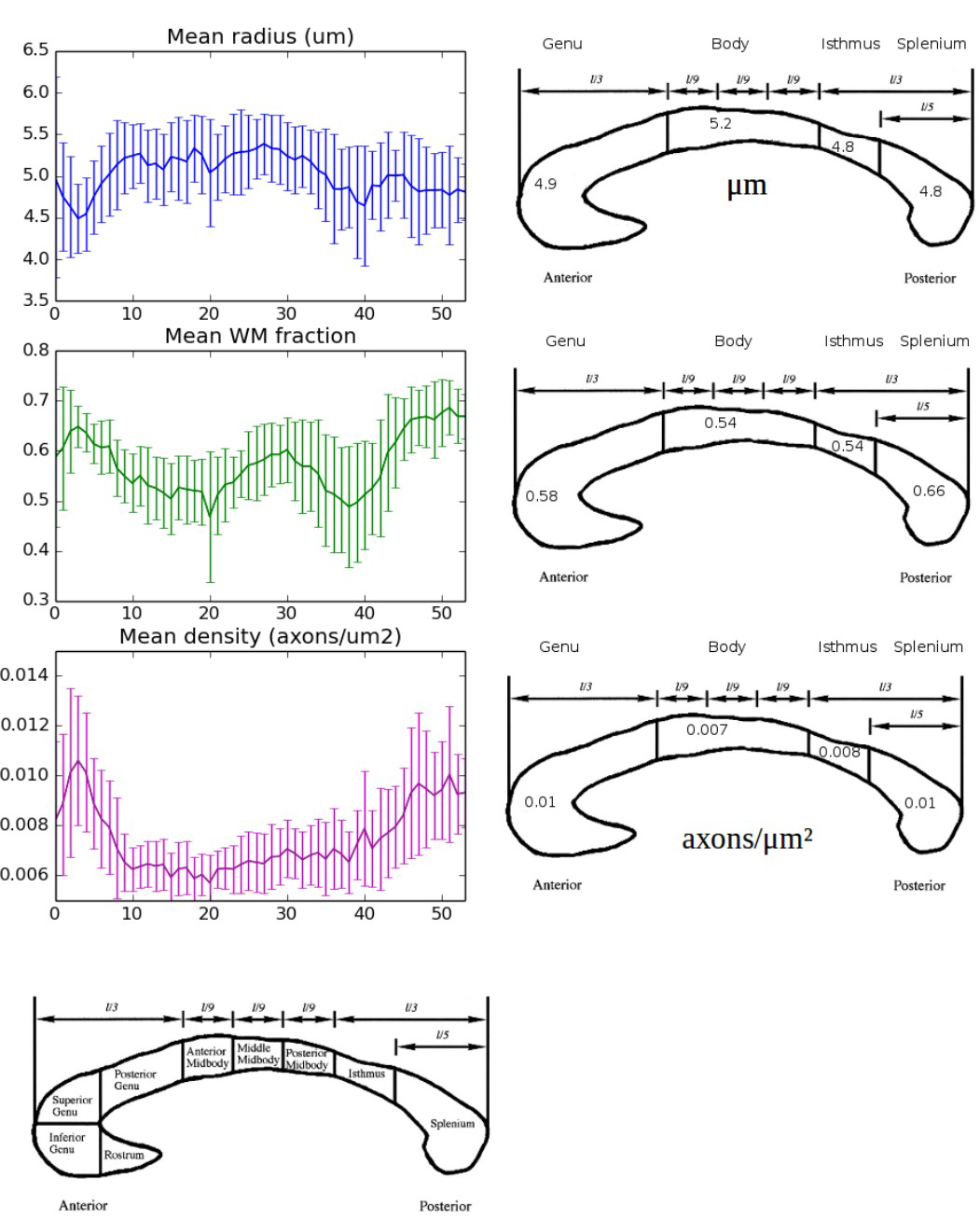
Deux protocoles ont été validés à 7T reposant pour l’un sur un schéma de type simple Pulsed Gradient Spin Echo doté de gradients trapézoidaux et a permis de cartographier finement la cytoarchitecture du corps calleux de 10 volontaires sains, comme l’illustre la figure 5 (le corps calleux a été choisi comme faisceau test, puisque la direction de ces fibres est connue d'emblée et peut donc amener à une simplication du modèle en faisant l'a priori de l'orientation initiale des fibres qui le composent). En tout point du corps calleux sont fournis la densité axonale locale, le diamètre axonal moyen ainsi que la dispersion angulaire moyenne. On retrouve bien le profil attendu de ces paramètres le long du plan sagittal médian, à savoir une densité axonale plus élevée et un diamètre axonal plus faible au niveau du genou et du splenium du corps calleux, et une densité axonale plus faible et un diamètre axonal plus élevé au niveau de son corps. On retrouve également une dispersion angulaire plus faible au niveau de la partie centrale du corps calleux qu’au niveau du splénium et du genou. Ce travail démontre l’énorme potentiel de la microscopie IRM de diffusion pour le développement de nouveaux biomarqueurs des pathologies cérébrales, donnant une information beaucoup plus pertinente sur les mécanismes physiopathologique en jeu qu’au travers de l’étude de paramètres scalaires tels que le coefficient de diffusion apparent (ADC), les diffusivités longitudinales ou transversale ou l’anisotropie fractionnelle (FA) issus des modèles n’exploitant que le profil angulaire du signal pondéré en diffusion, et ne permettant généralement pas de comprendre et caractériser de manière fine les modifications du tissu s’opérant à l’échelle cellulaire au cours du développement de la pathologie.
Dans le cadre du consortium européen Human Brain Project (https://www.humanbrainproject.eu) auquel elle appartient et ayant pour objectif de simuler le fonctionnement du cerveau humain, l'équipe METRIC travaille en étroite collaboration avec l'UNATI et l'UNIACT, dans le cadre du sous-project SP2 mené en partenariat avec de nombreux acteurs européens (et plus particulièrement l'équipe des Pr Katrin Amunt et Karl Zilles du Forschungszentrum Jülich), et a pour mission de construire in vivo le premier atlas quantitatif de la l'ultrastrucure de la substance blanche et de la cytoarchitecture du cortex cérébral, grâce aux méthodes de microscopie par IRM de diffusion.