La membrane biologique est organisée en deux feuillets de lipides dans lesquels des protéines sont partiellement ou totalement enchâssées. La composition lipidique des deux feuillets est différente, ce qui permet de contrôler de nombreux processus biologiques (trafic membranaire, signalisation cellulaire…). L’asymétrie des bicouches lipidiques résulte de nombreux mécanismes dont les principaux contributeurs sont les protéines transmembranaires de transport de lipides. Les floppases catalysent le transport de lipides du feuillet interne (cytosolique) vers le feuillet externe (exoplasmique) des membranes et les flippases font le travail inverse. Chez l’Homme, des mutations de plusieurs homologues des protéines flippases sont impliquées dans des formes rares de cholestase intrahépatique ou d’affection neurologique (syndrome d’ataxie cérébelleuse associé à une déficience intellectuelle). Des travaux menés chez le rongeur montrent leur implication dans la sécrétion de l’insuline, la forme des globules rouges ou encore la survie des photorécepteurs de la rétine. Chez les plantes, les flippases sont essentielles à la réponse au stress thermique. Des efforts importants sont déployés pour déterminer les structures de ces protéines et éclairer ainsi les mécanismes de transport des lipides. À cet égard, les flippases intriguent. Elles constituent le seul sous-type (P4) de la famille des ATPases de type P à transporter des lipides. Les autres sous-types, P1 à P3, transportent uniquement des ions, des molécules environ dix fois plus petites que les lipides (le substrat du sous-type P5 n’est pas connu). Comment en adoptant une même structure globale[1], les ATPases de type P4 ont-elles pu évoluer par rapport aux autres sous-types et créer en leur sein un « chemin » suffisamment large pour faire passer un substrat « géant » ?
Des équipes du SB2SM/I2BC (Université Paris-Sud/CEA/CNRS), de l’Université d’Aarhus et de l’Institut Max Planck à Francfort se sont associées pour déterminer la structure de la protéine Drs2 en complexe avec sa sous-unité associée Cdc50, une flippase de la levure. Drs2 contient un segment à son extrémité C-terminale qui permet l’auto-inhibition de son activité. Cette auto-inhibition est partiellement levée en présence de phosphatidylinositol-4-phosphate (PI4P)[2] ; on parle alors de conformation intermédiaire. La troncation de l’extrémité C-terminale, en présence de PI4P, rend l’enzyme pleinement active. Les équipes ont obtenu la structure du complexe Drs2-Cdc50 dans chacune de ces trois conformations : auto-inhibée, intermédiaire et active. Cette première pour la famille des flippases a pu se faire grâce à la cryo-microscopie électronique (technique d’imagerie qui a valu le prix Nobel de chimie à trois chercheurs en 2017). Les trois structures, obtenues à des résolutions de 2,8 à 3,7 Å, révèlent le mécanisme par lequel l’extrémité C-terminale de Drs2 inhibe l’activité du complexe et les premières étapes de la levée de cette auto-inhibition par le PI4P (figure 1). La comparaison des structures révèle également l’existence d’une cavité par laquelle les chercheurs proposent que la tête polaire du lipide substrat (la phosphatidylsérine principalement) soit prise en charge lors de son cheminement d’un feuillet à l’autre des membranes cellulaires (figure 2).
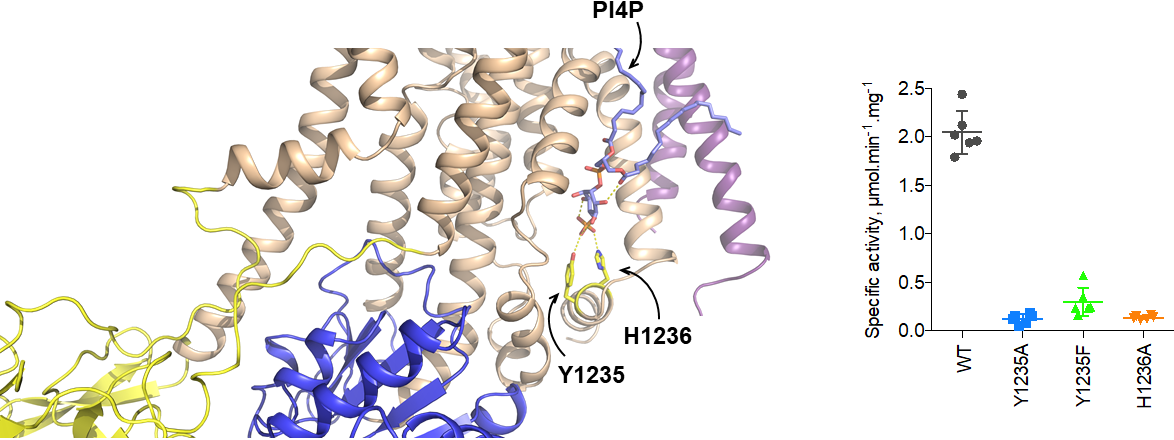
|
|
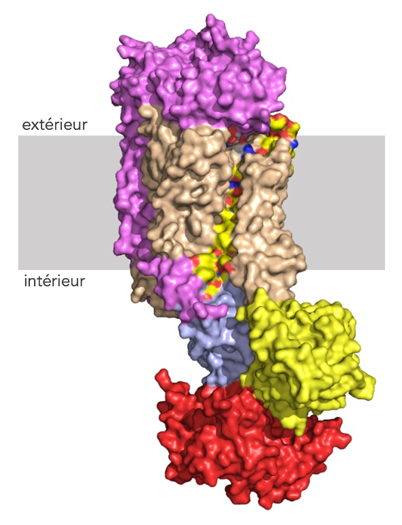
Voie possible de transport du lipide à travers la bicouche lipidique. L'hélice transmembranaire 4 de Drs2, qui borde la cavité, est représentée en jaune, le reste du domaine transmembranaire en brun. Les domaines cytosoliques A, N et P de Drs2 sont respectivement colorés en jaune, rouge et bleu clair. La sous-unité Cdc50 est représentée en rose. © G. Lenoir/Université Paris-Saclay
|
Ces travaux décrivent pour la première fois l’architecture moléculaire d’une flippase mais d’autres structures d’ATPases de type P4, en présence de leur substrat lipidique et/ou de partenaires régulateurs, devront être obtenues pour élucider totalement le mécanisme de « flip » des lipides et voir si celui-ci est conservé d’un eucaryote à l’autre.
Cette publication a fait l'objet d'un communiqué de presse.
[1] La présence de nombreux résidus conservés entre les différents sous-types est en faveur d’un mécanisme catalytique commun. De plus, les structures à haute résolution de membres des sous-types P1, P2 et P3 déjà connues indiquent que l’architecture serait conservée pour l’ensemble de la famille.
[2] Le phosphatidylinositol-4-phosphate (PI4P) est un phospholipide abondant dans les membranes de l’appareil du Golgi, organite intracellulaire important pour le trafic vésiculaire et la maturation des protéines.