Chez les organismes eucaryotes multicellulaires, l'ADN génomique se réplique de manière contrôlée dans le temps en ordonnant des régions de réplication précoces, intermédiaires ou tardives selon un programme de réplication spatio-temporel. La manière dont ce programme est orchestré demeure mal comprise, mais il a été observé que son dérèglement entraînait une instabilité génomique souvent observée dans le cancer. La protéine Rif1 est un régulateur clé du programme de réplication chez les eucaryotes mais son rôle au cours des premiers cycles cellulaires embryonnaires, lorsque la réplication de l'ADN est très rapide et que les facteurs de réplication sont abondants, reste mal caractérisé.
Dans cette étude, les auteurs ont utilisé un système in vitro très performant d'extraits d'œufs de Xénope qui mime les premiers stades de développement précoce. Ils ont démontré, par une approche de peignage moléculaire de l'ADN, que la déplétion de Rif1 dans ce système conduisait à une augmentation substantielle de l'activation des origines de réplication à l'échelle de groupes d'origines. D'autres observations, combinées avec un modèle numérique, suggèrent que la déplétion de Rif1 accélère le programme de réplication sans en modifier les paramètres locaux, comme les distances entre les origines voisines sur les fibres d'ADN. En d'autres termes, la déplétion de Rif1 conduirait à un programme plus homogène et temporellement compressé. Le modèle numérique a également prédit que Rif1 pourrait influencer la disponibilité de facteurs limitants de la réplication. Une prédiction vérifiée expérimentalement qui montre que l'absence de Rif1 s'accompagne effectivement d'un recrutement chromatinien accru d'une protéine kinase de la phase S (phase du cycle cellulaire dans laquelle l'ADN est répliqué) et de plusieurs autres facteurs clés impliqués dans l'initiation de la réplication.
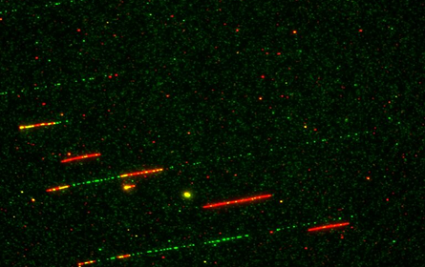
Fibres d'ADN peignées (vert) avec régions répliquées (rouge) en l'absence de Rif1 © Kathrin Marheineke / I2BC
En proposant un modèle dans lequel ils montrent in-vitro et in-silico que la protéine Rif1 restreint simultanément l'accès à l'ADN ou l'activité de plusieurs facteurs pour affiner la synthèse d'ADN exceptionnellement rapide observée au cours du développement embryonnaire, les auteurs de la présente étude ouvrent la voie vers l'élucidation des mécanismes moléculaires impliqués dans certaines maladies résultant de mutations ou de variants de Rif1 chez l'homme.
Texte repris de l'actualité de l'I2BC. Pour en savoir plus, lisez la suite dans Nature Portfolio
Contacts : Kathrin Marheineke (kathrin.marheineke@i2bc.paris-saclay.fr) ; Arach Goldar (arach.goldar@cea.fr)
Le peignage moléculaire de l'ADN est une technique utilisée pour étirer les molécules d'ADN de manière uniforme et parallèle et peut être appliqué à des études de la dynamique de la réplication de l'ADN.