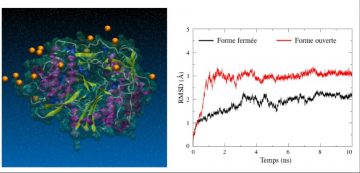
L’étude du régulateur transcriptionnel Slr1738 de Synechocystis est impliqué dans la tolérance de cette cyanobactérie aux métaux lourds et au stress oxydant. Il a en effet été montré que la souche mutante D1738 est plus résistante que la souche sauvage à un stress oxydant provoqué par H2O2 mais aussi beaucoup plus sensible que cette dernière lorsqu’elle est exposée à un stress métallique. L'équipe a développé une nouvelle méthode pour la construction du complexe SLR1738 avec son ADN cible basée sur les éléments de structure secondaire. Le modèle tridimensionnel ainsi et que les différentes formes qui en découlent permettent d'expliquer les propriétés biologiques remarquables de ce facteur de transcription (formes altérées après oxydation ou glutathionylation,switch biologique pour la transcription).
IRM du xénon et biosensing
Le xénon 129 hyperpolarisé obtenu par pompage optique à l'aide de lasers est utilisé en RMN afin de servir soit de sonde d'environnement (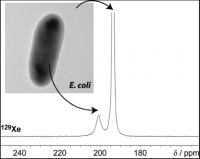 gamme de déplacement chimique très importante ~250ppm) soit d'agent de contraste pour faire de l'imagerie par résonance magnétique. Ces travaux sont menés en collaboration avec le laboratoire de Patrick Berthault (Laboratoire de Structure et Dynamique par Résonance Magnétique) du CEA/DSM Saclay. Concernant l'interactions du xénon avec des cellules, nous avons démontré que le spectre du xénon nous renseigne sur 1) l'état de la cellule (morte ou vivante) 2) l'écart de déplacement chimique entre le signal intracellulaire et extracellulaire est directement indicateur du type de cellule observé, signature cellulaire.
gamme de déplacement chimique très importante ~250ppm) soit d'agent de contraste pour faire de l'imagerie par résonance magnétique. Ces travaux sont menés en collaboration avec le laboratoire de Patrick Berthault (Laboratoire de Structure et Dynamique par Résonance Magnétique) du CEA/DSM Saclay. Concernant l'interactions du xénon avec des cellules, nous avons démontré que le spectre du xénon nous renseigne sur 1) l'état de la cellule (morte ou vivante) 2) l'écart de déplacement chimique entre le signal intracellulaire et extracellulaire est directement indicateur du type de cellule observé, signature cellulaire.
Interaction protéines-nanoparticules
L'utilisation croissante des nanoparticules (NPs) soulève des inquiétudes quant à leur éventuelle toxicité. Alors que de nombreux travaux étudient ces questions toxicologiques in vivo ou au niveau cellulaire, nous proposons une démarche orientée avant tout vers une compréhension mécanistique des interactions protéines-NPs. Les objectifs sont de mieux comprendre les mécanismes moléculaires d'adsorption des protéines sur les NPs, de déterminer si les protéines adsorbées ont des caractéristiques physico-chimiques, structurales ou fonctionnelles particulières, et le cas échéant de développer des algorithmes de prédiction d’adsorption sur les NPs. A partir de ces connaissances, des perspectives fonctionnelles sont attendues, notamment en terme toxicologique, telles que la prédiction et l'identification de cibles cellulaires et enzymatiques privilégiées. Ce projet transversal (DSV-DSM) nécessite des compétences techniques variées et une forte complémentarité entre les équipes (biochimistes, physico-chimistes, bioinformaticiens et structuralistes).
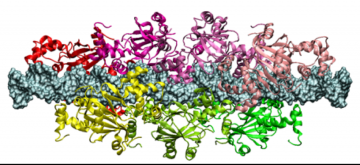
Polymérase du virus de l'hépatite C
Chez les virus à ARN positif comme le virus de l’hépatite C, l’activité mais aussi la fidélité de la polymérase sont finement régulées. Ceci permet d’une part la synthèse d’ARN viral uniquement au bon endroit et au bon moment, d’autre part le maintien de la diversité de la quasi-espèce virale qui infecte un hôte donné. L’étude de ces phénomènes à l’échelle atomique permet d’une part d’en avoir une compréhension fine, d’autre part d’améliorer les inhibiteurs antipolymérase. Nous proposons de combiner l’expertise structurale de différents groupes afin de répondre à deux questions centrales de l’activation de la polymérase (passage à la forme ouverte) et de sa fidélité (incorporation). Les études expérimentales seront guidées et complémentées par des approches computationnelles.